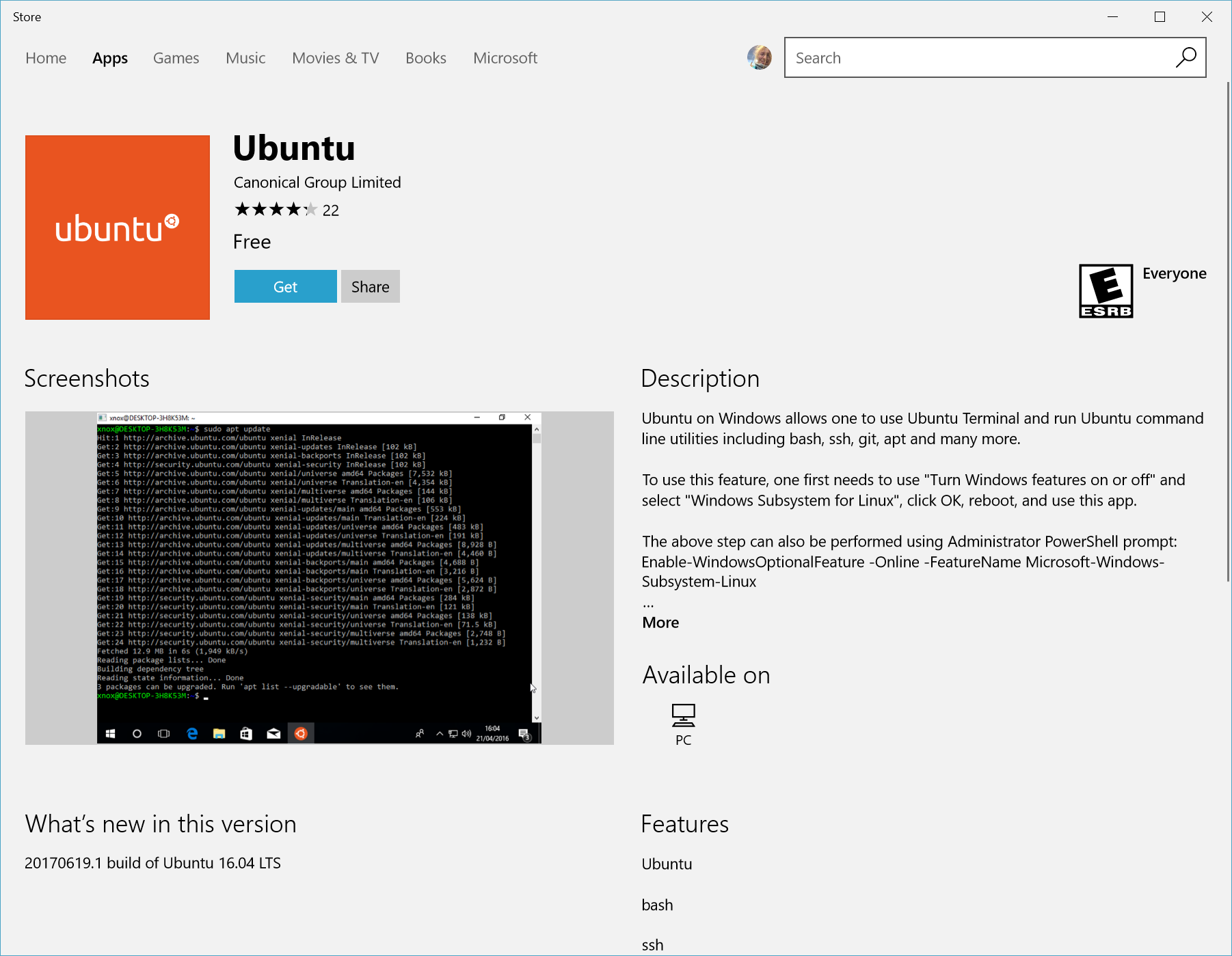
Bioinformática Use Case
A partir da versão 10+ do MS Windows é possível utilizar o Ubuntu através do Subsistema Windows para Linux (WSL), como se o Ubuntu fosse uma aplicação instalada dentro do Windows.
Fonte: https://docs.microsoft.com/pt-br/windows/wsl/install-win10
A virtualização na BIOS já foi ativada.
Abra o PowerShell como administrador (na barra de busca do windows digite PowerShell e clique com o botão direito e selecionar Executar como administrado):
dism.exe /online /enable-feature /featurename:Microsoft-Windows-Subsystem-Linux /all /norestartPara verificar a sua versão e o número de build, selecione a tecla do logotipo do Windows + R, digite winver e selecione OK
Antes de instalar o WSL 2, você precisa habilitar o recurso opcional Plataforma de Máquina Virtual. No PowerShell como administrador execute:
dism.exe /online /enable-feature /featurename:VirtualMachinePlatform /all /norestartBaixe e instale o pacote mais recente (x64):
Depois que a instalação for concluída, vá para a próxima etapa: configurar o WSL 2 como a sua versão padrão ao instalar novas distribuições do Linux. No PowerShell como administrador execute:
wsl --set-default-version 2Abra a Microsoft Store e escolha sua distribuição do Linux favorita, no nosso caso o Ubuntu.
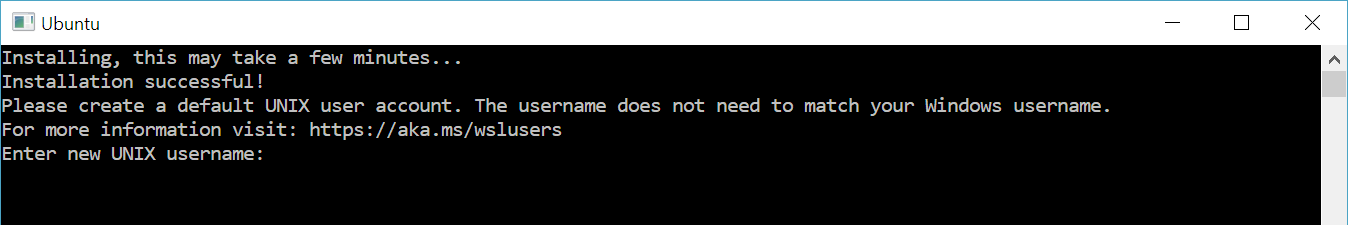
Na primeira vez que você iniciar uma distribuição do Linux recém-instalada, uma janela de console será aberta e será solicitado que você aguarde um ou dois minutos para que os arquivos sejam descompactados e armazenados em seu PC. Todas as futuras inicializações deverão levar menos de um segundo.
Em seguida, você precisará criar uma conta de usuário e uma senha para sua nova distribuição do Linux.
O C: do seu Windows é montado como uma partição dentro do Ubuntu e fica em /mnt/c
O c:\Users\puga que é seu HOME no Windows não é o mesmo do Ubuntu
# comando listar as partições existentes
# df: comando que lista as partições
# -h: listar de forma que humano entenda (MB, GB ou TB)
df -h
....O C: fica /mnt/c
# entrando no C: do seu Windows
# no caso da Puga é puga
cd /mnt/c/pugaPuga decidiu criar um diretório na Área de Trabalho (Desktop) chamado bioinfo:
# entrando o diretorio Desktop
cd /mnt/c/puga/Desktop
# mkdir: criando o diretorio bioinfo
mkdir bioinfo
# entrando no diretório bioinfo
cd bioinfo# estrutura de diretorios
# app: coloque aqui os programas instalados (fora do apt-get install)
# bam: coloque os arquivos .bam e .bai
# instalacao: arquivos de instalacao .exe ou .gz que não quer deletar
# docs: suas documentacoes ou helps
mkdir app bam instalacao docsVamos atulizar as bibliotecas no sistema para pode instalar os softwares de bioinformática.
sudo: é o comando para acionar os poderes de super usuário (como por exemplo para instalar coisas)
# atualiza a lista de repositórios
sudo apt-get update
# atualiza os programas mais novos
sudo apt-get upgradePodemos executar em um comando a instalação dos programas juntos
sudo apt-get install -y bwa samtools freebayesSolitice direto no site do Annovar http://download.openbioinformatics.org/annovar_download_form.php e se seu e-mail estiver na whitelist você receberá o link para download da versão mais nova.
Salve no seu diretório app. Agora, acesse o terminal do Ubuntu, vá até o diretório app e digite os comandos:
# tar -zxvf
# avisos: pode aparecer alguns avisos de conflito (ignore)
# entrar no diretorio app
cd /mnt/c/puga/Desktop/bioinfo/app/annovar
# listar conteudo do diretorio
ls -1
annovar.tar.gz
# descompactar
tar -zxvf annovar.tar.gz
# entrar no diretorio
cd annovar
# listar conteudo do diretorio
ls -1 Lista dos bancos de dados disponíveis para o Annovar: https://annovar.openbioinformatics.org/en/latest/user-guide/download/.
Verificar sempre se existe novas versões para os bancos de dados
# entrar no diretorio do annovar
cd /mnt/c/puga/Desktop/bioinfo/app/annovar
# clinvar
perl annotate_variation.pl -buildver hg19 -downdb -webfrom annovar clinvar_20200316 humandb/
# gnomad
perl annotate_variation.pl -buildver hg19 -downdb -webfrom annovar gnomad211_genome humandb/Bucket do Google Cloud para as referências do Broad Institute
Nesse bucket do Google/Broad tem todas as referências e os indexação dessa versão do genoma do BWA (Homo_sapiens_assembly19.fasta.64.*). Todas as referências tem um tamanho de ~30GB.
- gcp-public-data--broad-references/hg19/v0
Para não travar o computador, principalmente quando trabalhamos com arquivo grandes, podemos dividir a chamada de variantes por cromossomos e no final podemos juntar os arquivos .vcf em um único para cada amostra ou um único para todas as amostras. Vamos fazer um teste com o chr22 na amostra CP8.bam:
- Clique no link e salve os arquivos
references_hg19_chr22.faereferences_hg19_chr22.fa.faino diretório/mnt/c/puga/Desktop/bioinfo/referencia
# entrar no diretorio bioinfo
cd /mnt/c/puga/Desktop/bioinfo/
# rodar freebayes apenas para o chr22 (Ver opções)
freebayes -f reference/references_hg19_chr22.fa -F 0.01 -C 1 --pooled-continuous --region 22 bam/CP8.bam > CP8.chr22.vcf# entrar no diretorio bioinfo
cd /mnt/c/puga/Desktop/bioinfo/
# anotar apenas com RefSeq
perl app/annovar/table_annovar.pl --vcfinput CP8.chr22.vcf app/annovar/humandb/ -buildver hg19 -out CP8.chr22 -remove -protocol refGene -operation g -nastring .# visualizar com less
# -S: linha única sem quebra
# -N: números no começo das linhas
# Para sair aperte a tecla 'q'
less -SN CP8.chr22.hg19_multianno.txt